Апробация мультиплексного SSR-анализа для ДНК-паспортизации сортов риса

Использование молекулярных ДНК-маркеров открывает большие перспективы для детального картирования хромосом, идентификации генов, их клонирования и конструирования новых сортов растений. С введением молекулярных маркеров в практику биологических исследований появились новые возможности изучения генетического разнообразия, определения родства на внутрии межвидовом уровне. Многочисленными… Читать ещё >
Апробация мультиплексного SSR-анализа для ДНК-паспортизации сортов риса (реферат, курсовая, диплом, контрольная)
Использование молекулярных ДНК-маркеров открывает большие перспективы для детального картирования хромосом, идентификации генов, их клонирования и конструирования новых сортов растений. С введением молекулярных маркеров в практику биологических исследований появились новые возможности изучения генетического разнообразия, определения родства на внутрии межвидовом уровне. Многочисленными исследованиями было показано, что одна из наиболее информативных ДНК-систем молекулярного маркирования сельскохозяйственных культур — так называемые микросателлитные последовательности ДНК (SSR). В геномы эукариот сплошь встроены простые последовательности, которые могут состоять из 4, 3, 2 и даже одного нуклеотида. Позже эти регионы были названы «микросателлитами» [1, 2].
Источник полиморфизма этих последовательностей — в сайт-специфическом варьировании длины повтора, что в свою очередь обусловлено различием в числе единиц повтора. Эксперименты по изучению генетического разнообразия культурного риса обнаружили до 25 аллелей на один микросателлитный локус. У риса наиболее часто встречаются динуклеотидные повторы типа (АТ)n, (GA)n, (GT)n, причем AT-повторы являются наиболее полиморфными и могут быть использованы для определения степени генетического родства между близкими по происхождению сортами [3], среди тринуклеотидных повторов преобладают (GGT)n [4], из тетрануклеотидных повторов следует отметить (GATA)n — как наиболее распространенный в геноме риса [5].
В результате работ Akagi et al (1996) и McCouch et al (1997) при скрининге геномной библиотеки риса общее количество микросателлитов было оценено в 5700−10 000, причем относительная частота встречаемости различных повторов уменьшается с увеличением размера повтора [6, 7].
Последующее секвенирование генома риса позволило предположить, что общее количество микросателлитных последовательностей, составляет порядка 100 000 [5].
С использованием микросателлитных ДНК-маркеров в мире выполнен широкий перечень исследований по оценке генетического разнообразия и ДНК-паспортизации сортов. Так, в работе авторского коллектива из Индии была выполнена оценка уровня полиморфизма 50 SSR-маркеров на выборке из 24 сортов риса подвида Индика. При этом, на основе полученных данных, не только отобрали наиболее полиморфные ДНК-маркеры для дальнейшего изучения индийского генофонда, но и оценили степень генетического родства изученных сортов и составили их ДНК-паспорта [8].
В другом исследовании, выполненном международным научным коллективом из Южной Кореи и Китая, было выполнено генотипирование 150 сортов риса, представляющих генетические ресурсы Южной Кореи, Китая и Японии, использовали выборку из 28 SSR-маркеров для оценки генетических дистанций изученных групп генотипов и сравнительного анализа уровня генетического разнообразия внутри групп. Было выявлено, что наибольшим уровнем аллельного полиморфизма по изученным локусам обладает генплазма риса из Южной Кореи. Наименьшим уровнем аллельного разнообразия обладала выборка генотипов из Японии. Общий уровень генетического разнообразия внутри групп южнокорейских и китайских сортов риса был выше, чем в выборке сортов из Японии [9]. Наряду с анализом генетической структуры коллекций сортов риса, SSR-маркеры также успешно применяются и для изучения генетического разнообразия природных популяций дикорастущих предковых видов риса [10], а также для анализа генетических взаимосвязей между современными сортами, стародавними сортами народной селекции и дикорастущими формами [11, 12].
Очевидно, что SSR-маркеры являются эффективной ДНК — маркерной системой для выполнения широкого спектра исследований, направленных на анализ генетического разнообразия коллекций генофонда риса, изучение филогенетических взаимосвязей на уровнях подвид/вид/род. В связи с этим в задачу работы входило формирование и апробация мультиплексных наборов SSR-маркеров, позволяющих эффективно проводить генотипирование сортов риса. Разработанные наборы были успешно использованы для SSR-паспортизации ряда сортов риса из коллекции генетических ресурсов ВНИИриса.
Материал и методы исследований
Образцы ДНК выделяли из свежесрезанной части листовой пластинки гибридных растений на различных стадиях вегетационного развития. Экстракцию ДНК проводили буфером следующего состава: 20мл 1 М Tris-HCl (pH 7.5), 5 мл 5MNaCl, 5 мл 0.5MEDTA (pH 8.0), 5 мл 10% SDS в общем объеме 100 мл. Часть листа (2−3см) растирали в 500 мкл экстрагирующего буфера в пластиковой пробирке объемом 1,5мл. Инкубировали образцы при 600С в течение 3 часов. Отделяли супернатант центрифугированием при 12 000 об/мин. К перенесенной в чистую пробирку верхней фазе добавляли 500 мкл изопропанола, оставляли для преципитации на 10−20 минут при комнатной температуре, предварительно перемешав. После этого образец центрифугировали 5 минут при 12 000 об/мин, полученный осадок промывали 300мкл 70% этанола, высушивали и растворяли в 200мкл 0,1*ТЕ. В ПЦР смесь добавляли по 2,5 мкл раствора ДНК, выделенного данным методом.
ПЦР проводили по стандартным методикам, но с выполнением предварительной оптимизации ряда параметров реакции [13]. Предварительную оценку продуктов ПЦР проводили электрофорезом в 2% агарозном 30−50 минут при напряжении 150V. В качестве красителя использовали бромистый этидий; гелевые пластины визуализировали в ультрафиолете.
Анализ размеров амплифицированных фрагментов SSR — маркеров проводили на автоматическом генетическом анализаторе ABIprism 3130. Обработку данных осуществляли в программе GeneMapper 4.1. Фрагментный анализ SSR-маркеров был выполнен на оборудовании ЦКП «Геномные и постгеномные технологии» ФГБНУ СКЗНИИСиВ.
При объединении ДНК-маркеров в мультиплексные учитывали размеры амплифицируемых фрагментов, температуру отжига праймеров, степень самокомплементарности праймеров, вносимых в одну смесь.
Результаты
В ходе выполнения исследований, на основании анализа литературных данных были отобраны 14 SSR-маркеров, из которых на данный момент апробированы 7 маркеров: RM1, RM11, RM70, RM122, RM164, RM167, RM168. Маркеры объединены в два мультиплексных набора, позволяющих проводить генетический анализ одновременно по 3 и 4 микросателлитным локусам одновременно. При этом SSR-маркеры, входящие в один мультиплексный набор, имеют неперекрывающиеся диапазоны размеров амплифицированных фрагментов. Это обеспечивает избегать ошибочной идентификации при детекции размеров ампликонов на автоматическом генетическом анализаторе ABIprism 3130. Для каждого из маркеров в мультиплексном наборе был использован специфичный для него флуоресцентный краситель: карбоксифлуоресцеин (FAM), 6-карбоксиродамин (R6G), карбокси-Х-родамин (ROX), тетраметилкарбоксиродамин (TAMRA). Каждый из приведенных красителей имеет разные оптические спектры флуоресценции, что позволяет проводить в одной реакции одновременную идентификацию нескольких фрагментов, синтезированных с использованием праймеров, меченых разными красителями. В таблице 1 представлены разработанные мультиплексные наборы.
Таблица 1 Мультиплексные наборы SSR-маркеров.
MLPX1. | MLPX2. |
RM1 (TAMRA). RM11 (ROX). RM70 (R6G). RM122 (FAM). | RM164 (TAMRA). RM167 (ROX). RM168 (R6G). |
При формировании комбинаций SSR-маркеров и последующей апробации мультиплексных наборов проводили оптимизацию следующих экспериментальных параметров:
- — соотношение концентраций праймеров ДНК-маркеров, входящих в один мультиплексный набор;
- — количество циклов реакции, температура отжига праймеров при постановке ПЦР: 30, 35, 40 циклов реакции; температура отжига праймеров: 52, 56, 58, 62єС;
Главной задачей при оптимизации параметров ПЦР было получение максимального синтеза целевых фрагментов при минимальном синтезе неспецифических амплификатов. Кроме того, с целью сокращения затрат времени при выполнении генотипирования больших объемов образцов и упрощения алгоритма работы, задачей также являлась разработка одногоунифицированного протокола ПЦР для всех мультиплексных наборов. В результате, для использованных в работе SSR-маркеров была принята наиболее оптимальная программа ПЦР:
- 1-й цикл: начальная денатурация — 5 минут при 94 °C;
- 2-этап: 35 циклов:
- 30 секунд денатурация при 94 °C;
- 30 секунд отжиг праймеров при 56 °C;
- 30 секунд синтез при 72 °C;
последний цикл синтеза 5 минут при 72 °C.
Оптимальной концентраций для всех используемых праймеров в ПЦР — смеси была определена концентрация 0,3 мМ. Концентрация дезоксинуклеотидтрифосфатов составила 0,05 мМ. Оптимальные условия ПЦР позволили получить в ходе фрагментного анализа на автоматическом генетическом анализаторе ABI prism 3130 достоверно идентифицируемые целевые фрагменты (Рисунок 1).
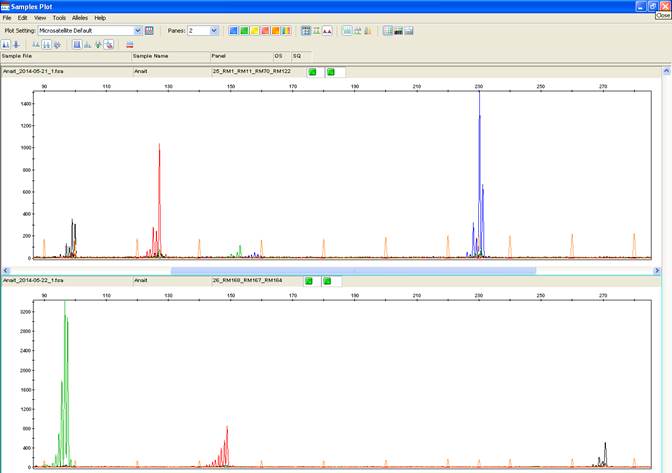
Рисунок — Результаты фрагментного анализа сорта риса Анаит по мультиплексным наборам SSR-маркеров. 1- MLPX1, 2 — MLPX2.
Как видно из результатов фрагментного анализа с использованием автоматического генетического анализатора ABIprism 3130 (Результаты представлены в рабочем окне программы Gene Mapper 4.1), оптимальные экспериментальные параметры и сочетания SSR-маркеров в мультиплексных наборах позволяют безошибочно идентифицировать целевые пики электрофореграмм. Для каждого SSR-маркера в мультиплексном наборе характерен пик со специфичным цветом на электрофореграмме. Это позволяет проводить достоверную идентификацию целевых фрагментов.
С использованием разработанных мультиплексных наборов было выполнено генотипирование некоторых отечественных и зарубежных сортов риса из коллекции генетических ресурсов ВНИИриса. Для всех изученных сортов были составлены SSR-фингерпринты, которые включают информацию об идентифицированных аллелях у каждого конкретного сорта (таблица 2).
Таблица 2 SSRфингерпринты сортов риса
RM1. | RM11. | RM70. | RM122. | RM168. | RM167. | RM164. | |
Олимп. | 100*. | ||||||
Визит. | |||||||
Дружный. | |||||||
Павловский. | |||||||
Приморский 1. | |||||||
Приморский 29. | |||||||
Южанин. | |||||||
Полевик. | |||||||
Боярин. | |||||||
ИР — 36. | |||||||
ВНИИР 9751. | |||||||
ВНИИР 9556. | |||||||
Диамант. | |||||||
Каприз. |
*Примечание: размер аллелей указан в парах нуклеотидов.
Как видно, все изученные сорта риса, представленные в таблице, имеют специфичный ДНК-профиль. Один из маркеров (RM168) у отечественных сортов имеет низкий уровень полиморфизма — один аллель 97 п.н. Однако, при этом, у сорта ИР-36, селекции Международного научно-исследовательского института риса (Филиппины) по данному локусу идентифицируется второй тип аллеля 107 п.н. Кроме того, по локусам RM1, RM11, RM167 и RM164 данный сорт имеет уникальные аллели, не представленные ни у одного из отечественных сортов. Это согласуется со значительными генетическими различиями данного сорта и остальных сортов из изученной выборки.
Апробированные в ходе работы SSR маркеры и сформированные из них мультиплексные наборы могут быть эффективно использованы для выполнения дальнейших исследований по ДНК-паспортизации и изучению генетического разнообразия отечественных коллекций генетических ресурсов данной культуры.
мультиплекс рис генотипирование.
- 1. Конарев А. В. Использование молекулярных маркеров в работе с генетическими ресурсами растений / А. В Конарев // Сельскохозяйственная биология, 1998. № 5. С.3−25.
- 2. Хавкин Э. Е. Молекулярные маркеры в растениеводстве / Э. Е Хавкин // Сельскохозяйственная биология, 1997. № 5. С.3−19.
- 3. Akagi H. Highly polymorphic microsatellites of rice consist of AT repeats, and classification of closely related cultivars with these microsatellite loci / H. Akagi, Y. Yokozeki, A. Inagaki, T. Fujimura // Theor. Appl. Genet., 1997. V. 94. P.61−67.
- 4. Wu K.S. Abundance, polymorphism and genetic mapping of microsatellites in rice/ K.S. Wu, S.D. Tanksley // Mol. Gen. Genet., 1993.-V.241. P. 225−235.
- 5. McCouch S.R. Microsatellite markers in rice: abundance, diversity, and applications/ S.R. McCouch, S. Temnykh, A. Lukashova, et al. // Rice genetic 4. Proceeding of the fourth international rice genetic symposium.- Los Banos., 2001. P. 117−135.
- 6. Akagi H. Microsatellite DNA markers for rice chromosomes / H. Akagi, Y. Yokozeki, A. Inagaki, T. Fujimura // Theor. Appl. Genet., 1996. V. 93. P. 1071−1077.
- 7. McCouch S.R. Microsatellite marker development, mapping and applications in rice genetics and breeding / S.R. McCouch, X. Chen, O. Panaud., et al. // Plant Mol. Biol., 1997. — V.35. P. 89−99.
- 8. Priyanka S. Allelic Diversity Among Basmati and Non-Basmati Long-grain Indica Rice Varieties using Microsatellite Markers / S. Priyanka, J. Sunita, S. Navinder, et al. // Plant Biochemistry & Biotechnology, 2004. V. 13. P. 25−32.
- 9. Weiguo Z., Jong-Wook C., Kyung-Ho Ma et al. Analysis of Genetic Diversity and Population Structure of Rice Cultivars from Korea, China and Japan using SSR Markers/ Z. Weiguo, C. Jong-Wook, Kyung-Ho Ma. et al // Genes and Genomics, 2009. V. 31. № 4. P. 283−292.
- 10. L.-Z.Gao. Comparisons of microsatellite variability and population genetic structure of two endangered wild rice species, Oryza ru? pogon and O. of? cinalis, and their conservation implications/ L.-Z.Gao, C.-H. Zhang // Biodiversity and Conservation, 2005. V. 14. — P. 1663−1679.
11. Michael J. Thomson. Genetic diversity analysis of traditional and improved Indonesian rice (Oryza sativa L.) germplasm using microsatellite markers/ Michael J. Thomson, Endang M. Septiningsih, Fatimah Suwardjo. et al. // Theor Appl Genet, 2007. V. 114. P. 559−568.
- 12. Basabdatta Das. Genetic diversity and population structure of rice landraces from Eastern and North Eastern States of India/ Basabdatta Das, Samik Sengupta, Swarup K. Parida //BMC Genetics, 2013. V. 14. P. 71.
- 13. Шибата Д. К. Полимеразная цепная реакция и молекулярно-генетический анализ биоптатов // Молекулярная клиническая диагностика.- М.: Мир, 1999. С. 395−427.